Las funciones básicas de los seres vivos las realizan las proteínas, cuya síntesis está dirigida y controlada por los ácidos nucleicos. Éstos contienen el "prospecto", las instrucciones de un organismo a partir de sus proteínas.
Los ácidos nucleicos poseen un programa, cuando se ejecuta da lugar a un ser vivo capaz de nutrirse, relacionarse y producirse.
Los ácidos nucleicos son macromoléculas formadas por la unión de muchos monómeros, denominados nucleótidos. Hay dos tipos:
○ ADN, ácido desoxirribonucleico
○ ARN, ácido ribonucleico
Nucleótidos, enlaces y funciones:
Los nucleótidos son las unidades que forman los ácidos nucleicos. Cada uno está formado por tres moléculas:
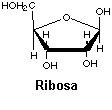
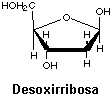
○ Pentosa → B-D-Ribofuranosa (ARN), B-D-Desoxirribofuranosa (ADN)
○ Ácido ortofosfórico → H3PO4
○ Bases nitrogenadas, son las que diferencian a un nucleótido de otro
-Púrica → adenina, guanina
-Pirimidínica → timina, citosina, uracilo
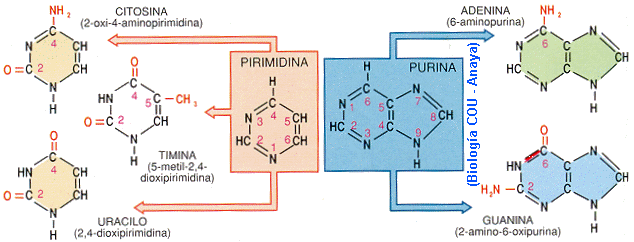
Los dobles enlaces son resonantes (cambian de posición), lo que hace que haya muchos electrones deslocalizados, es decir, libres, produciendo cargas fluctuantes que les permite a las bases nitrogenadas capturar protones (H+); característica exclusiva de las bases del nitrógeno.
Las bases orgánicas son de nitrógeno. El -OH del carbono 1 de la pentosa con la base púrica o base pirimidínica forma un enlace glucosílico (entre dos bases), el enlace carece de puente de oxígeno.El -OH del carbono 5 de la pentosa se une con un -OH del ácido fosfórico formando un enlace éster. Todo el conjunto se llama nucleótido 5' monofosfato.
Unidos a otras moléculas forman compuestos de gran importancia biológica.
Compuestos derivados de los nucleótidos no nucleicos:
○ Coenzimas:
- AMP → adenosin monofosfato
- ADP → adenosin difosfato
- ATP → adenosin trifosfato

Forman un enlace de alta energía por la repulsión eléctrica del fósforo.
Estos enlaces sirven para almacenar energía. Se rompen fácilmente por hidrólisis y liberan la energía que necesitaron para formarse, por eso, funcionan como enzimas transportadores de energía.
La energía química, de enlace, es la única forma de energía utilizable por los seres vivos.
Coenzimas transportadores de energía : ATP / UTP / GTP / CTP
○ AMPc: (adenosin monofosfato cíclico)

Es el nucleótido de adenina que en el carbono 5' tiene un ácido fosfórico que forma un segundo enlace con el carbono 3' de la adenina. El enlace es un enlace éster intramolecular.
El AMPc se conoce como el segundo mensajero. Es un mediador entre moléculas extracelulares portadoras de información, como ; las neuronas y los neurotransmisores, en el interior de la célula es donde tiene su efecto.
Es el responsable de la respuesta celular a la hormona. La hormona es el primer mensajero.
○ Nucleótidos 5' más otras moléculas:
Forman: NAD+, NADP+, FMN+, FAD+
Todos son coenzimas redox, transportadores de electrones y protones.
Enlace fosfodiester o enlace nucleósido:
Es un tipo de enlace covalente que se produce entre un grupo hidroxilo (-OH) en el carbono 3' y un grupo fosfato (H3PO4) en el carbono 5' del nucleótido entrante, formandose así un doble enlace éster.
De esta forma se unen cientos y miles de nucleótidos.
Por convenio se dice que el primer nucleótido de una cadena es aquel que tiene el carbono 3' libre. El último sería el carbono 5' combinado con el fósforo (fosforilado).
Las cadenas van en dirección 3' → 5'.

Estructura de los ácidos nucleicos:
Del ADN:
Es un polímero lineal de desoxirribonucleótidos 5' MP de adenina, timina, guanina y citosina.
Cuando se pone en medio acuoso espontáneamente (igual que las proteínas) toma un estado nativo , su forma espacial (tridimensional) es hasta cuatro niveles estructurales (primaria, secundaria, terciaria, cuaternaria), cada uno depende del anterior.

Estructura primaria:
Es la unión de los nucleótidos mediante enlaces fosfodiéster. Dado que los nucleótidos son diferentes (A, T, G, C) tienen información genética, orden, secuencia de nucleótidos.
AATCCCGG ATCGCGCT
La secuencia es específica en cada especie y específica de cada individuo.
La información genética es igual a la secuencia de nucleótidos, igual que los planos para construir las proteínas.
Estructura secundaria:
Se descubrió de una forma indirecta y rocambolesca un químico llamado Chargaff que realizó un análisis de ADN.
A G púricas
▬ = ▬ = ▬▬▬▬▬ = 1
T C pirimidínicas
Dos biologas Franklin y Wilkin hicieron radiografías del ADN. Descubrieron que el ADN era helicoidal y fibroso.
Watson y Crick eran investigadores, descubrieron el modelo de la doble hélice. Este modelo explicaba los datos de Chargaff, los rayos x y sus propiedades.
Según su modelo, el ADN forma dos cadenas en hélice enfrentadas por las bases y unidas entre sí por puentes de hidrógeno, igual que una escalera de caracol en la que los pasamanos serian las pentosas y fósforos y los peladaños las bases enfrentadas y unidas por puentes de hidrógeno.
Según su modelo es una hélice a derechas, las cadenas son antiparalelas.
3' → 5'
5' ← 3'
Las dos cadenas están trenzadas, para separar las cadenas hay que destrenzarlas. Son complementarias, siempre va unida A-T, G-C, lo sacaron de Chargaff, se forma el máximo número de puentes de hidrógeno que le dan estabilidad a la hélice.
El diámetro de la cadena es de 20 Ã (Amstrong), siempre se une una base púrica con una base pirimidínica.
Las cadenas están unidas por puentes de hidrógeno que son débiles, explica la fácil desnaturalización y renaturalización del ADN.
Estructura tericaria y cuaternaria:
Se debe resolver el problema de como meter cadenas con 1.000.000 de nucleótidos en un espacio tan reducido (célula).
Es diferente en los procariotas y en los eucariotas:
○ Procariotas, tienen 0.003 mm de longitud. Lo consiguen superenrollando la cadena de ADN, la cierran sobre sí igual que un cromosoma circular. Todo ello sin ayuda de las proteínas.
○ Eucariotas, su tamaño es 1.000.000 veces mayor que las procariotas. El ADN tiene 2 metros de longitud, dentro de un núcleo que tiene 0.000000002 m. de diámetro. Lo consiguen con la ayuda de las proteínas, la doble hélice da dos vueltas sobre un cilindro de proteínas, llamado nucleosoma y el conjunto forma un collar de perlas.
Se enrolla sobre sí mismo y da lugar al selenoide, éste se pliega y repliega formando bucles, se le conoce como cromatina (forma activa del ADN).
Cuando se va a efectuar la división, los bucles se compactan y empaquetan, se hace visible, esto es la cromatida.
De los ARNs:
Son polímeros lineales de ribonucleótidos 5' MP (ribosa) de adenina, guanina, citosina o uracilo (cambia timina), también pirimidínica.
Tienen más cargas negativas (por 1.000), las cadenas son más inestables, se rompen más fácilmente.
La estructura de los ARNs es monocatenaria, pero complementaria consigo misma, formando puentes de hidrógeno intracatenarios, doble hélice en la estructura secundaria. La parte no complementaria forma las discontinuidades, son los bucles.
Esto da lugar a la estructura terciaria, en forma de hoja de trébol, es el ARN transferente.
ARN mensajero:
La estructura primaria tiene una relación lineal con la estructura del ADN, son complementarias.
El ARNm copia la información genética del ADN, su código genético, y lo transmite a la célula, lo lleva hasta los ribosomas encargados de fabricar las proteínas.
El ARnm tiene unos pocos minutos de vida antes de ser degradado, es lo óptimo, de otro modo la proteína se fabricaría indefinidamente.
Como todos los ARNs se fabrican en el núcleo a partir del ADN y realizan su función en el citoplasma.
Entre 3-5% del ARN total lo constituye el ARNm. Sólo tiene estructura primaria.

ARN ribosómico:
Constituye entre 80-85% del ARN total.
Con estructura primaria y partes complementarias donde forma doble hélice (estructura secundaria) y discontinuidades que en conjunto forman la estructura terciaria.
Los distintos ARNr se combinan con 70 proteínas para formar los ribosomas, que es el único orgánulo macizo que poseen las células, cuya finción es leer el código genético del ARNm y fabricar la proteína expresada en ese gen.
Los ribosomas leen y traducen el código genético.
ARN transferente:
Son el 10% del total de los ARNs celulares.
Son pequeñas moléculas que tienen un 80% de bases complementarias dentro de su cadena, entre los que se forman doble hélice (estructura secundaria), más bucles, boomerang u hoja de trébol.
Son específicos de aminoácidos (ARNt, Aa) que se unen por su extremo 3' a una adenina (siempre ACC), también son específicos de anticodón (triplete complementario del codón correspondiente de ese aminoácido).
Se forman en el núcleo a partir del ADN y pasan al citoplasma donde realizan su función que es transportar su Aa hasta los ribosomas donde son ensamblados en el orden indicado en el ARNm, el mismo que se encuentra en el gen del ADN.

Funciones biológicas de los ácidos nucleicos (genética molecular):
ADN: el código genético:
El ADN se divide en fragmentos, llanmados genes, estos genes contienen la información genética para fabricar las proteínas (como planos para construir las proteínas).
La mayoría de genes sirven para fabricar proteínas, los ARNm y otros genes sirven para los ARNt, ARNr.
El ADN contiene el código genético (información genética).
El código genético es un idioma, formado por letras que son A, T, C, G.
Con 20 Aa se pueden formar combinaciones con repetición de 4,3 = 64 palabras distintas, igual triplete/codón.
Un gen dice todo lo que tiene que decir para formar una proteína completa.
La unión de los Aa forma la cadena de proteínas completa.
Gen significa también ARNt, ARNm.
En células procariotas los genes son continuos. En células eucariotas son discontinuos.
Hay partes que tienen sentido (exones) y partes sin sentido (intrones) que no sirven para fabricar proteínas, tienen función estructural.
Los exones tienen información para fabricar las proteínas.
La mutación se produce en los intrones. La maduración de los ARNm, elimina a los intrones.
Características del código genético:
○Universal, es igual para todos los seres vivos (desde virus hasta nosotros), todos procedemos de un antecesor común.
○No-solapado, una base solamente pertenece a un triplete.
○No tiene ni puntos ni comas, tiene señales de iniciación, empieza por AUG y termina en UGA, UAA, UAG.
○Degenerado, un mismo Aa está determinado para varios codones. Un mismo Aa se puede decir de varias formas distintas.
Leucina → CUC / CUG / CUU / CUA
○No es imperfecto, sería imperfecto cuando un codón tuviera varios aminoácidos.
Es degenerado para facilitar la síntasis de proteínas.
La complementariedad codón / anticodón sólo afecta a las dos primeras bases, la tercera se tambalea, vacila, lo que facilita la llegada y salida de los ARNt (como debe ser), a los ribososmas durante la síntesis de proteínas.
Replicación del ADN:
La replicación es el proceso por el cual el ADN se copia para poder ser transmitido a nuevos individuos.
El ADN contiene la información genética, cada vez que una célula se divida debe formarse una nueva copia idéntica para repartira entre las dos células hijas, antes de dividirse.
En 1953, Watson y Crick propusieron el mecanismo de duplicación de las cadenas de ADN. En principio había tres posibles modelos de replicación:
○Modelo semiconservativo, se obtienen dos moléculas de ADN nuevas, formadas ambas por una hebra original y una hebra nueva.
○Modelo conservativo, las cadenas iniciales se conservan y se forman dos cadenas nuevas.
○Modelo dispersivo, las cadenas iniciales se fragmentan y aparecen en las cadenas nuevas.
La duplicación ocurre al final de la interfase, fase S (ciclo celular, son las fases por las que pasa una célula en su vida).
Ciclo celular:
Antes de entrar en división la célula se duplica en la fase S.
Básicamente el ADN polimerasa utiliza como molde las cadenas del ADN para formar nuevas cadenas complementarias.
La duplicación la realizan, ADN polimerasa I y ADN polimerasa III.
Son las más complejas de todas las enzimas que hay en la naturaleza.
ADN polimerasa I, tiene varios centros activos, uno de ellos se agarra a la cadena molde, otra localiza -OH libre del carbono 3' del último nucleótido que colocó en la cadena de formación. Por otro se une al nuevo nucleótido que va a incorporar a la cadena, es trisfosforilado y complementario con el molde (si es así forma un enlace éster 5'←3'). Tiene actividad exonucleasica (significa que mira atrás).
Si comete un error, no complementario con el molde, lo corta y rellena el hueco correctamente.
El proceso de duplicación es semejante en procariotas y eucariotas.
Comienza la duplicación por el origen de la replicación, se separan las cadenas, se forma las cadenas complementarias y progresa la duplicación como una horquilla de replicación. Ésta progresa como una cremallera al abrirse.
La ADN polimerasa le ocurre lo mismo que a los ARN polimerasa sólo pueden utilizar como orden la cadena 3'→5'.
La cadena se duplica de una forma continua y la hebra retardada se duplica a trozos de 3'→5', su síntesis es discontinua, es más lenta.
La duplicación progresa en las dos direcciones.
A diferencia de lo que pasa con ARN polimerasa, "saben" comenzar una cadena.
Las ADN polimerasa "no saben", sólo continuan una cadena ya empezada, así la duplicación comienza con un ARN cebador, el primer sintetizado por la primasa (ARN polimerasa), que luego continua la ADN polimerasa III.
Luego se corta el primer y se rellena el hueco dejado por el ARN cebador, realizado por ADN polimerasa I. Se unen los fragmentos (llamados de Okazaki, son la unión de un ADn con un ARN) mediante una enzima llamada ligasa una vez en la hebra conductora y muchas veces en la hebra retardada.
En la duplicación intervienen otras proteínas no enzimáticas, como la helicasa que rompe los puentes de hidrógeno del ADN para separar las cadenas.
La topoisomerasa relaja el desenrollamiento de las cadenas al estar enrolladas.
La SSB es una proteína que estabiliza la cadena (molde) sencilla del ADN para que no vuelvan a unirse.
La duplicación es compleja y rápida, se polimerizan 45•10(3) nucleótidos por minuto. Es muy precisa, sin errores, comete un error por cada 1 / 10-100•10(6) nucleótidos polimerizados.
Cuando hay un error la ADN polimerasa I lo corta y rellena el hueco y luego la ligasa repara los errores.
No todos los errores son reparados (1 / 10(4)•10(6) nucleótidos polimerizados).
Los errores que no se reparan son mutaciones.
Si la mutación no compromete la viabilidad es beneficiosa para el individuo, sino es beneficiosa aumenta la variabilidad genética.
Cuando hay mutaciones aparecen individuos diferentes.
Sin mutación en la selección natural mueren todos y con mutación, adaptación, evolución ante un medio cambiante.
La selección natural elige de lo que hay.
Transcripción:
La transcripción es el proceso de copia de un gen o fragmento de ADN utilizando ribonucléotidos y originándose diferentes tipos de ARN.
La estructura primaria del ADN determina la estructura primaria de la proteina. Se podría hacer directamente pero no se hace porque el ADN es enorme, están en el núcleo, los ribosomas están en el citoplasma.
Se forman un intermediario, el ARNm que lleva la información de la estructura primaria del ADN, del núcleo hasta los ribosomas.
La cadena de ADN se copia en ARNm y éste pasa a la proteína.
La síntesis del ARNm es la transcripción (por extensión se llama síntesis a todos los ARNs).
La transcripción consiste en la síntesis de un ARN complementario. A / U, G / C, C / G, T / A.
La transcripción la realizan las enzimas, ARN polimerasa con ADN dependiente.
Los ARN polimerasas se fijan a ciertas promotoras del ADN, donde hay muchas timinas y adeninas, son distintas en procariotas y eucariotas.
Desenrollan una vuelta de hélice y utiliza una cadena de ADN como molde para formar una cadena de ARN complementaria.
Al avanzar desenrolla otra vuelta de hélice y la anterior se vuelve a enrollar, así funciona hasta que llega al final del gen.
Al final del gen se desprende la ARN polimerasa.
La transcripción es semejante en los procariotas y eucariotas, con una diferencia, los genes de los procariotas son continuos, el ARNm no necesita maduración, puede ser directamente traducido por los ribosomas.
En los eucariotas los genes son discontinuos.
Se transcribe todos los exones e intrones.
El ARNm no puede ser traducido directamente, necesita maduración para eliminar los intrones.
La realizan las ribonucleoproteínas pequeñas nucleares (enzimas RNPpn).
Las RNPpn están formadas de proteínas y de ARN, el ARN es complementario con las secuencias iniciales y finales de los intrones, a los que se une cortándolos.
Luego se pegan los exones, lo realiza una enzima llamada ligasa (ADN polimerasa).
La ADN polimerasa se fija en la cadena 5'←3' como molde, fabrican una cadena de ARNm complementaria al ADN y antiparalela.
Después de añadidos unos 30 ribonucleótidos al extremo 5' se le pone una caperuza de 7-metilguanosina trifosforilada. Esto se hace para que el ribosoma reconozca el comienzo del gen.
Tanto en procariotas como en eucariotas al final del gen hay TTATTT, eso el el final de la transcripción.
En el 3' del ARNm se añaden 200 ribonucleótidos de adenina, eso se conoce como cola poli A.
En los procariotas sólo hay un ARN polimerasa que depende del ADN.
En los eucariotas hay tres:
─ARN polimerasa I, sirve para la síntesis de ARNr
─ARN polimerasa II, sirve para la síntesis de ARNm
─ARN polimerasa III, sirve para la síntesis de ARNt
Traducción en procariotas:
Previamente se debe transcribir su gen en forma de ARNm (transcripción) por parte de la ADN polimerasa II que utiliza como molde la cadena 3'→5' del ADN y forma un ARNm que es complementario y antiparalelo (primer nucleótido, el 5' libre) a ella, esto ocurre en el citoplasma.
También previamente los Aa deben ser activados, a los que se le añade energía para que puedan formar enlaces peptídicos, covalentes. Activación Aa+energía+ARNt
Su ARNt suyo específico que está unido en el código genético al anticodón.
La aminoacil ARNt sintetasa, coge el Aa, el ARNt, el ATP y 2 H2O y lo pasa a:
Aa+ARNt+ATP+2 H2O → ARNt aminoacil+AMP+2 Pi+H2O
Utiliza la energía para formar el enlace éster. ARNt▬O▬Aa, se produce en el extremo 3' donde siempre hay una adenina. 2 fósforos para formar el enlace éster entre el ARNt y el Aa.
Ribosomas:
Los ribosomas son cada uno de los orgánulos de las células vivas, compuestos de ácido ribonucleico y proteínas, que se ocupan de la síntesis de estas últimas.
Los procariotas son 70s (coeficiente de sedimentación),tienen dos subunidades, cada subunidad tiene su propio coeficiente de sedimentación, el mayor 50s y el menor 30s.
Los eucariotas son 80s, el mayor 60s y el menor 40s.
Los ribosomas a nivel molecular son complejos supramoleculares, están formados de varios ARNr (65% más 50 proteínas diferentes).
Las dos subunidades se fabrican por separado en el núcleo, salen al citoplasma, autoensamblan por esteroespecificidad en torno a 1 ARNm (sin ARNm se sparan no hay síntesis de proteínas).
Aparecen propiedades nuevas en el ribosoma:
○Capacidad de interaccionar reversiblemente con otras moléculas, ARNt aminoacil o con unas proteínas que se llaman factores, que llegan y salen del ribosoma provocando cambios estructurales en cada molécula del ribosoma y en el ribosoma completo. Estos cambios desembocan en la síntesis de proteínas. Básicamente los ribosomas favorecen el acceso de los ARNt aminoacil del ARNm (coloca los Aa en su orden), leen el mensaje y forman enlaces peptídicos entre sus Aa, es decir, traducen.
○Hidrólisis de GTP, interaccionan en la síntesis de proteínas, tiene tres fases:
-Iniciación
-Elongación
-Terminación
Iniciación:
La subunidad 30s del ribosoma se coloca en el 5' del ARNm en los dos primeros dobletes, el primero es AUG, codón iniciación.
El AUG es el codón correspondiente al anticodón UAC que lleva el Aa llamado formil metionina.
El formil metionina es un Aa metionina aminobloqueado para que el formilo no pueda formar enlace peptídico.
En los eucariotas empiezan por metionina.
Luego llega la subunidad 50s y ya está formado el complejo de iniciación.
Varios factores de iniciación entran y salen del ribosoma y se hidroliza un GTP.
Elongación:
Tiene tres fases:
○Fijación:
Llega el segundo triplete y se coloca el codón ante el anticodón.
Dos factores de elongación y la hidrólisis permite la fijación.
○Formación del enlace peptídico:
El enlace peptídico lo forma la enzima peptidil transferasa en 50s.
Transfiere la energía del enlace éster ARNt aminoacil para formar el enlace peptídico entre los aminoácidos.
○Translocación:
Para que ocurra la translocación tiene que llegar otro factor de elongación (diferente a la fijación), más hidrólisis de GTP. El ribosoma se desplaza un triplete en dirección 3'. Sale por degenerado.
Se llama sitio P de peptidil al que tiene el ARNt con el péptido en formación.
Sellama sitio A de aminoacil en donde se coloca un aminoacil ARNt.
Una nueva fijación en el sitio A, un enlace peptídico y otra translocación hasta que se llega a la terminación.
Terminación:
Habría una cadena de formación en le sitio P hasta la translocación y se encuentra el anticodón de terminación (UGA, UAA, UAG).
Este anticodón se encuentra bloqueado por factor de terminación.
El sitio P transfiere la energía del enlace éster al agua porque no hay ARNt.
Se produce hidrólisis del enlace éster, se libera la cadena peptídica por la hidrólisis y se separan las subunidades del ribosoma.
En procariotas este proceso tan complejo monta 1400 Aa por minuto tras lo cual se degrada el ARNm, sino se formaría constantemente la misma proteína.
Traducción en eucariotas:
Al principio de cada proteína hay metionina.
En procariotas los ribosomas están aislados.
En eucariotas los ribosomas están reunidos. Se llaman polisomas, que son varias cadenas, se forman al mismo tiempo.
En eucariotas hay ribosomas que están asociados a la membrana y ribosomas libres.
En procariotas los ribosomas están libres.
Los ribosomas se encuentran asociados a la membrana nuclear o a la membrana del retículo endoplasmático rugoso.
Las proteínas se introducen en el núcleo o retículo endoplasmático rugosos.
El que tiene el poli A hace que la transcripción se realice en la membrana tanto nuclear como la del retículo endoplasmático rugoso. Se pega a la membrana a través de un receptor.
La transcripción empieza por el 5', empieza con ribosomas libres.
Todos los ARNm transcritos a nivel de membrana comienzan por una secuencia de Aa apolares (secuencia hidrófoba o señal).
Cuando se acerca a la membrana atrae a ciertas proteínas intrínsecas de la membrana (apolares) que difunden en torno a la secuencia señal por apolares.
Cuando las proteínas intrínsecas tocan la secuencia hidrófoba, se disuelve la secuencia señal en las proteínas intrínsecas, introduciéndose a través de la membrana igual que en un túnel.
La secuencia señal tiene como función permitir la entrada de la proteína en formación a través de la membrana.
Detrás entra toda la proteína en formación independientemente de que sea hidrófoba porque la subunidad 60s estabiliza el túnel.
En ese momento se corta la secuancia señal y queda la verdadera proteína, el túnel desaparece.
Regulación de la expresión génica:
Un gen es el fragmento más pequeño de una molécula de ADN que posee información completa para un carácter determinado.
Para regular un carácter determinado hay que regular el funcionamiento de las proteínas por parámetros generales:
○Temperatura, pH, concentración de sustrato, concentración de coenzimas, son inespecíficos, afectan a todas las proteínas por igual.
○Activación / inhibición enzimática.
○Enzimas reguladores (alostéricos y modulados covalentemente).
○Ribosomas, modificando la velocidad de traducción.
○Modificaciones post-traducción, la pepsina en vez de fabricarla, fabrica pepsinógeno inactivo que es pepsina inactiva.
Son formas derrochadoras del control de la transcripción.
El control de la transcripción permite economizar y adaptarse a las condiciones cambiantes del medio.
En procariotas y eucariotas existe el control de transcrpción.
En los eucariotas, además, hay un control de la transcripción de otro tipo que permite un proceso en la diferenciación celular. Las células se diferencian en su estructura y funcionamiento.
La estructura y el funcionamiento van determinadas por las proteínas que poseen.
Todas las células poseen el total de la información genética, podrían fabricar todas las proteínas del organismo (embrionarias, células madres)(son totipotentes).
La diferenciación celular se consigue por la represión irreversible del 90% de su genoma en las primeras fases del desarrollo embrionario.
La heterocromatina (se tiñe), ADN empaquetado, inactivo, reprimido irreversiblemente.
La eurocromatina (no se tiñe), bucles, ADN activo, formando proteínas.
El control reversible de la transcripción existe en procariotas y eucariotas, es semejante pero en eucariotas es más complejo.
La presentan todas las procariotas por estar en contacto con un medio cambiante.
También lo presentan algunas células eucariotas, glóbulos blancos, células del hígado, del riñon, del epitelio (tubo digestivo).
El resto no, como neurona, muscular, etc. porque el medio es estable, no es cambiante, medio interno (homeostasis).
Hay dos tipos de enzimas:
○Constitutivos, forman parte de la célula siempre, no tienen control de la transcripción, siempre son necesarios, reproducción celular, fotosíntesis, glucólisis.
○Inducibles, si tienen control de la transcripción, no siempre son necesarios.

Teoría del Operón:
Explica el control reversible de la transcripción.
Lo descubrieron Jacob y Monod (franceses), a partir de datos de otros investigadores.
Clarificaron las relaciones moleculares y genéticas durante la inducción / represión enzimática reversible, que sirve para cualquier proteína.
Con la escherichia coli (e. coli), otros investigaron que la galactosidasa es un enzima inducible reversiblemente (que formaba la e. coli).
Había una serie de mutantes en e. coli que:
- no formaban galactosidasa
- forma alterada
- constitutiva
Hicieron un mapa genético con los genes y sustancias responsables de la inducción / represión de la galactosidasa.
Los genes estructurales contienen la información para la síntesis de las enzimas que intervienen en la ruta metabólica. Son:
- gen "z", determina una enzima galactosidasa
- gen "y", determina una enzima permeasa
- gen "a", determina una enzima transacetilasa
Esta secuencia multienzimática actúa en la hidrólisis de la lactosa, sirve para cualquier proteína.
Los genes reguladores son los encargados de dirigir las sustancias represoras que se muestran en las actividades de los genes estructurales. Son:
- gen "i", gen inhibidor, forma la proteína represora activa, tiene dos centros activos, uno para el gen "o" y otro para el inductor (lactosa). Cuando está el inductor suelta el gen "o" si no está el inductor va unido al gen "o".
- gen "p", gen promotor, señal reconocida por la ARN polimerasa para comenzar la transcripción de los genes estructurales.
- gen "o", gen operador, a tope con la proteína represora, no deja avanzar a la polimerasa y por lo tanto no se produce la transcripción.
Los genes "p", "o", "z" se encuentran juntos, los otros genes pueden encontrarse en un lugar distinto.
Mutación en "z", se produce galactosidasa alterada.
Mutación en "p", no se forma galactosidasa.
Mutación en "o", galactosidasa como constitutiva-o (se fabrica siempre).
Mutación en "i", galactosidasa como constitutiva-i.
Si hay lactosa el gen "o" queda libre, se transcribe en "z", inducción.
Si no hay lactosa el gen "o" queda ocupado, no se transcibe en "z", represión.
Inducción / represión en una enzima o secuencia multienzimática por catabolito (sustancia del catabolismo).
Los genes estructurales relacionados entre sí forman secuencia multienzimática de la hidrólisis de la lactosa, regulados todos por los mismos genes reguladores.
Inducción / represión de una enzima o secuencia multienzimática por el producto final.
El gen inhibidor produce proteína represora inactiva más correpresor (producto final). Con producto final no se transcribe porque el gen "o" queda ocupado. Sin producto final el gen "o" queda libre y hay transcripción. Con producto final se vuelve la proteína represora activa, esto es conocido como retroalimentación negativa.
En la retroalimentación positiva si la bacteria come lactosa / glucosa (enzimas constitutivas), se come lo que puede diferir. El gen "p" junto a la proteína aceptora del AMPc y el AMPc, se transcribe, si falta alguna no se transcribe.